|
Благодаря методу визуализации, позволяющему получать изображение отдельных молекул, ученые Университета Иллинойса (University of Illinois) выявили механизм функционирования важного фермента, регулирующего процессы репарации ДНК.

Профессор физики Таекджип Ха (Taekjip Ha) и его аспирантка Джихае Парк (Jeehae Park использовали метод переноса энергии флуоресцентного резонанса для изучения того, как важный фермент ДНК регулирует репарацию ее поврежденных участков. Красный и зеленый флуоресцентные красители позволили им отследить функционирование отдельного фермента. (Credit: Photo by L. Brian Stauffer)
Ферменты хеликазы прежде всего известны тем, что расплетает двойную спираль ДНК в процессе репликации, но у них есть и другие функции, в частности, репарация ДНК. Ученые из Университета Иллинойса сфокусировали свое внимание на одной из бактериальных хеликаз – PcrA, принимающей участие в предотвращении нежелательной рекомбинации.
Молекулы ДНК состоят из двух цепочек, сплетенных в спираль. Если одна из цепочек повреждается или разрывается, окружающее пространство расплавляется, и молекула в этой области представлена только одной цепочкой. Сразу после повреждения специализированные белки начинают процесс рекомбинации – восстановления второй цепочки, используя неповрежденную цепочку ДНК в качестве шаблона.
«Процесс рекомбинации очень важен для репарации ДНК, но если он выходит из-под контроля, возникают проблемы», - говорит профессор физики Таекджип Ха (Taekjip Ha). «Хеликаза PcrA регулирует процесс рекомбинации, удаляя с цепочки ДНК рекомбинационные белки».
Используя метод переноса энергии флуоресцентного резонанса (fluorescence resonance energy transfer - FRET), Ха и его коллеги смогли установить один из механизмов, с помощью которого фермент PcrA регулирует процесс рекомбинации. Метод основан на изменении интенсивности свечения двух красителей-флуорофоров, зависящей от их близости друг к другу. Исследователи связали красители с противоположными концами «хвоста» одноцепочечной молекулы ДНК.
Хеликазы принадлежат к молекулам-моторам, то есть классу ферментов, использующих химическую энергию для перемещения вдоль молекулы ДНК. Но с помощью FRET ученые наблюдали многократно повторяющийся процесс, когда два красителя постепенно сближаются, а затем быстро удаляются друг от друга. Вместо перемещения вдоль одноцепочечного «хвоста» ДНК, PcrA связывается с ней в точке разрыва, где встречаются двух- и одноцепочечные участки. Затем хеликаза использует свою моторную функцию для подтягивания к себе конца одноцепочечной ДНК, подобно тому, как это проделывает рыбак со своей леской. Не перемещаясь вдоль одноцепочечной ДНК, фермент последовательно связывается с ее нуклеотидами, расположенными все ближе и ближе к концу цепочки (постепенное усиления свечения флуоресцентных меток).
«Сочетая структурно-специфическое связывание с ДНК и моторные функции, фермент «сматывает» ДНК, удаляя в процессе этого рекомбинационные белки», - говорит Ха, являющийся также ученым-исследователем Медицинского института Говарда Хьюза (Howard Hughes Medical Institute).
Когда фермент достигает конца цепочки, он отпускает его (быстрое ослабление свечения флуорофоров) и начинает процесс связывания сначала, удаляя любые сомнительные белки на поврежденной ДНК.
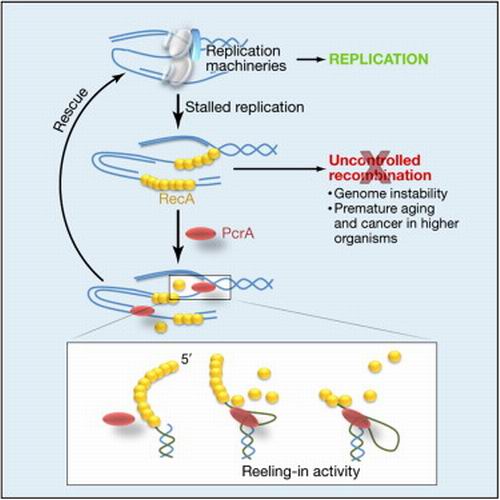 Транслокация ферментов хеликаз вдоль молекул нуклеиновых кислот лежит в основе ключевых функций клетки. Однако до сих пор не было ясно, как транслокация может приводить к удалению связанных с ДНК белков, и основные свойства, такие как элементарный размер шага, оставались спорными. Используя флуоресцентный анализ отдельных молекул хеликазы PcrA из Bacillus stearothermophilus, ученые из Университета Иллинойса установили, что PcrA скорее перемещается вдоль отдельной цепочки ДНК, чем расплетает шаблонную двойную спираль. PcrA прикрепляется к шаблонному дуплексу, используя субдомен 2В, и «подтягивает» к себе конец запаздывающей цепочки, образуя одноцепочечную петлю. Крайне часто повторяющееся образование петель выявляет, что PcrA перемещается с постоянным шагом размером в 1 нуклеотид. Эта активность требует открытой конформации PcrA и может быстро демонтировать ранее образованную нить RecA даже при низких концентрациях PcrA, обеспечивая механизм устранения потенциально вредных рекомбинационных интермедиатов. (Схема: сайт cell.com) Транслокация ферментов хеликаз вдоль молекул нуклеиновых кислот лежит в основе ключевых функций клетки. Однако до сих пор не было ясно, как транслокация может приводить к удалению связанных с ДНК белков, и основные свойства, такие как элементарный размер шага, оставались спорными. Используя флуоресцентный анализ отдельных молекул хеликазы PcrA из Bacillus stearothermophilus, ученые из Университета Иллинойса установили, что PcrA скорее перемещается вдоль отдельной цепочки ДНК, чем расплетает шаблонную двойную спираль. PcrA прикрепляется к шаблонному дуплексу, используя субдомен 2В, и «подтягивает» к себе конец запаздывающей цепочки, образуя одноцепочечную петлю. Крайне часто повторяющееся образование петель выявляет, что PcrA перемещается с постоянным шагом размером в 1 нуклеотид. Эта активность требует открытой конформации PcrA и может быстро демонтировать ранее образованную нить RecA даже при низких концентрациях PcrA, обеспечивая механизм устранения потенциально вредных рекомбинационных интермедиатов. (Схема: сайт cell.com)
С помощью разработанной Ха методики ученые смогли ответить и еще на один вопрос, касающийся PcrA: насколько постоянна моторная функция фермента. Ученые согласны с тем, что в среднем для перемещения на одну структурную единицу ДНК, то есть пару оснований, PcrA использует одну единицу клеточной энергии АТФ. Но так как традиционно фермент изучался на относительно больших образцах, сильный разброс данных привел к противоречивым взглядам (по некоторым данным до шести пар оснований на одну молекулу АТФ).
Так как метод FRET позволяет визуализировать отдельные молекулы, исследователи смогли проследить за этой функцией фермента и установили, что на перемещение на одну пару оснований PcrA действительно необходима одна молекула АТФ.
В ближайшем будущем ученые планируют создать реакционную среду, более соответствующую среде живой клетки, используя три и четыре красителя с разными цветами, чтобы одновременно измерить активность нескольких белков. Они также хотят понять, почему хеликаза перемещается только в одном направлении.
«Это идеальное сочетание новой технологии и интересной биологической проблемы», - считает Ха.
Результаты исследований опубликованы в журнале Cell.
По материалам
Related Articles: |













